细胞作为生命结构的基本单位,储存大量的生物信息。细胞异质性是生物组织的普遍特征,即使是相同类型的细胞受到周围微环境的影响也会存在基因表达的差异。细胞的发育是一个动态过程,处于不同阶段的同一细胞基因表达不同。以往的转录组测序技术(bulk RNA-sequencing,bulk RNA-seq)[3]需要对成千乃至上百万的细胞大样本进行分析,是对大量细胞测序分析得出的平均结果,或者反映的是数量占优势的细胞数据,这就会掩盖部分重要信息,忽略细胞之间或细胞亚型之间的异质性,不利于生物多样性的理解和研究。而对于干细胞、肿瘤细胞这类稀有细胞则因样本量较少,无法作出准确的数据分析。为了解单细胞异质性,近年来兴起的单细胞转录组测序技术(single-cell RNA-sequencing,sc RNA-seq)备受关注。
sc RNA-seq是从单个细胞水平获得全转录组的表达谱,其原理是将分离的单个细胞的微量全转录组RNA扩增后进行高通量测序。2009年,Tang等[4]将该技术应用于单个小鼠的卵裂球的基因分析中,发现了芯片未检测到的5200多个基因和1800个可变剪切点。单细胞测序文库构建流程主要包括单细胞提取、细胞裂解、mRNA 反转录、cDNA 扩增及文库构建等。本文将对sc RNA-seq过程中的几项基本技术特征进行分析,并阐述该技术在眼科研究领域中的应用。1sc RNA-seq的基本技术特征
1.1单细胞分离技术
目前,常用的单细胞分离技术主要包括显微操作(micromanipulation)、激光捕获显微切割(laser-capture microdissection,LCM)、荧光激活细胞分选(fluorescence-activated cell sorting,FACS)和微流控技术(microfluidics)等。因各项技术的灵敏度和产出率不同,应用范围亦有所不同。显微操作技术和LCM是依赖人工在高倍显微镜下观察细胞的形态和颜色特征来挑取单个细胞的方法,常用于从培养的细胞系、早期的胚胎细胞和固定组织切片中提取单个细胞。这两种方法要求操作者技术熟练,难度较大,耗时较长,产出率较低。FACS有20种不同的参数设置,可依据细胞大小和形态,或预先用荧光标记的抗体识别细胞表面标记物,通过流式细胞仪分选出目的细胞。FACS具有全自动和高通量的特点,但是该项技术需要细胞初始样本量较大,对于一些稀有细胞群体并不适用。微流控技术是在微米级通道中分离、捕获单个细胞,可以用于稀有细胞的筛选,该方法反应体系小、样本需求量小、节约试剂、减少污染,因此,微流控技术被认为是目前单细胞分离较好的方法。
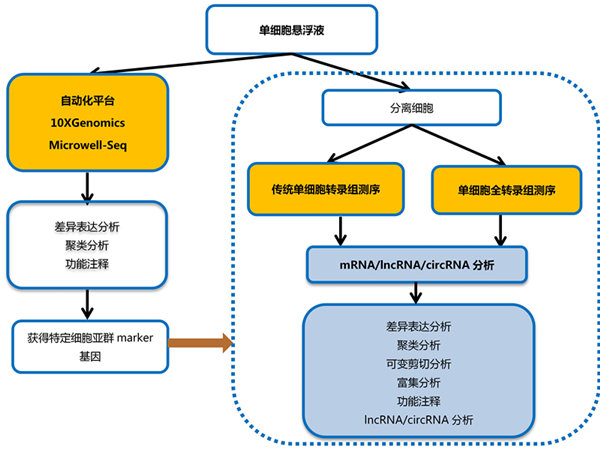
1.2 单细胞转录组的扩增及测序文库的构建
单细胞转录组的扩增是sc RNA-seq的关键步骤。根据合成第二条cDNA 链时使用的酶,可将单细胞转录组测序的cDNA 扩增方法分为PCR 法(如mRNA-Seq、Smart-Seq、Smart-Seq2、STRT-Seq和SMA)、体外转录法(如CEL-Seq) 和Phi29 聚合酶法(如PMA)。近年来,芯片液滴法作为新一代的文库构建方法,具有其独特的优势。
Islam等[11]发明的CytoSeq技术是将细胞悬液加入微反应孔中进行基因分析,用于研究细胞表达的差异,检测罕见细胞类型,该方法不受细胞体积的限制,可直接对不同大小和性状的混合细胞群进行研究。但该技术目前只能用于已知基因的研究,不能发现新基因。Drop-Seq[12]和inDrops[13]是将细胞悬液封装在含有DNA启动子条形码的水凝胶球的液滴中,然后细胞溶解,经反转录过程,mRNA被条形码标记。可追踪每个基因的细胞来源,显著提高了测序通量并降低了测序成本。液滴微流控平台可同时捕获上千个单细胞,并对其转录组或基因组进行测序,具有反应试剂少、效率高等特点。目前,许多平台已经商业化,并向研究人员提供单细胞转录组测序服务。Illumina是一个大量平行短序列测序平台[14],可同时对8个样本进行分析,细胞通量大,测序效率高,但仪器设备昂贵。
ChromiumTM Single Cell 3′Solution平台是10x Genomics公司研发的一种直接的、低成本的测序方法,一次可封装8个样本,耗时约6 min,细胞捕获效率近50%。利用一种新的简易算法“Supernova”,每次计算仅需2 d时间,大大提升了检测效率。目前已广泛应用于临床多个领域。ICELL8(WaferGen Biosystems)是一种新的单细胞RNA测序系统,芯片含有5184个反应孔,每个反应孔均填充有寡核苷酸编码的poly-d(T)、独特的条码和单一分子识别符,可捕获约1300个单个细胞。
该系统具有一个多样本纳米级分配器 Multi-Sample Nano-Dispenser (MSND),与显微镜连接,可在数分钟内对所有的反应孔进行观察,大约可以有37%的反应孔捕获单个细胞,MSND可以仅向含有单个细胞的反应孔中填加反应物,很大程度上节省了时间和费用。该系统可以从复杂样本中分离单个细胞,是一个高效经济的单细胞转录组测序方法,能够完成上千个细胞的表达谱分析,使得研究人员能够对复杂的生物样本细胞转录组进行破译,具有细胞重复率低、交叉污染少、纯度高等特点。Fluidigm公司研发的C1 单细胞扩增仪器[21],可以在同一芯片上自动完成Smart-seq步骤,将单细胞分离、反转录及cDNA扩增,构建cDNA文库,即可进行二代测序(next generation sequencing,NGS)或qPCR,显著提高了测序效率,省时省力。但是这些方法需要特殊仪器,耗资巨大。
而改良的Smart-seq2方法,可以同时对上百个细胞的转录组进行测序,不需要试剂盒或特殊的单细胞捕获工具。Gardeux的研究团队等开发了一个针对sc RNA-seq数据的完整分析的网络平台,即自动化单细胞分析途径(automated single-cell analysis pipeline,ASAP),运用这一平台,科研人员不需要具备强大的计算机技术和知识储备,仅使用各种常用的算法和工具来分析sc RNA-seq的数据。这为人们分析细胞多样性和异质性提供了有效的工具。Hu等在论文中描述了一种新的方法,即用机器学习算法[如support vector machine (SVM) 和random forest (RF)]分析sc RNA-seq数据。这是一种鉴别不同细胞中转录组数据差异的较佳方法,还可以扩展并适用于其他单细胞转录组测序表达谱的研究,如高度异质性肿瘤的进展和治疗。许多生物信息学工具是专门设计用于单细胞测序的数据分析,在干细胞领域被用来区分细胞类型及细胞亚型,确定标记基因。Monocle是近期发明的一种针对sc RNA-seq数据的无监督算法,通过对数据空间降维形成一个伪时间轨迹,对细胞发育和分化的进程进行时间序列重构。这种序列重排揭示了关键调控因子表达的闸门式变化,发现新型的细胞分化的调控因子。这项研究表明,用Monocle进行单细胞表达分析,揭示监管分化的新调控作用。
1.3 sc RNA-seq的应用
细胞是生物体的基本结构单位和功能单位,在其生长发育过程中,因细胞状态、环境刺激因素和内在程序的不同,转录组信息的变化呈多样性表现,这种多样性是构成细胞异质性的关键因素。
sc RNA-seq能够从单个细胞水平研究RNA的差异表达。自2009年,Tang等应用sc RNA-seq以来,这项技术已发展成为研究复杂生物体系细胞异质性的有效方法,被更多的研究人员应用于多个研究领域,包括干细胞发育和分化、胚胎期器官的发育、肿瘤、免疫、神经以及微生物等。
下面将对近年来该技术在眼科研究领域的应用进行综述。
2sc RNA-seq在眼科研究领域的应用
2.1sc RNA-seq在眼部细胞亚型及罕见细胞类型鉴定中的应用
神经系统是由多种细胞组成,仅与视觉相关的成年小鼠大脑视皮层就含有上百万个细胞。准确的细胞分类有助于科学家对大脑的发育、功能以及疾病状态等方面进行研究。近年来,根据sc RNA-seq在大脑皮层、下丘脑以及感觉神经元等方面研究的应用,将细胞按基因表达特征进行分类,使人们对神经多样性的理解更加深入。
视网膜属于中枢神经系统的一部分,含有60余种神经元,在视觉图像产生过程中起特定的作用。视觉图像的产生包括3个阶段,首先平行光线被光感受器细胞捕获,将光信号转变为电信号,然后经双极细胞传导至特定的神经节细胞形成神经冲动,经过视神经传入大脑视觉中枢。视网膜是一种易于观察、易于获取的组织,又具有一定的神经元多样性,是研究细胞发育、神经元分化以及神经系统多样性的模型,它由多种细胞类型组成,包括神经节细胞、小胶质细胞、无长突细胞、双极细胞、光感受器细胞以及视网膜色素上皮细胞等。对于如此复杂的组织结构,应用传统的大量细胞基因测序(bulk RNA-seq),将不能准确反映细胞之间的特异性。而sc RNA-seq则能够准确反映单个视网膜细胞的基因表达情况。
Goetz等在JoVE中阐述了单个视网膜细胞转录组基因表达分析的实验步骤。Laboissonniere等应用视网膜神经节细胞亚型进行分类,目的是将细胞的基因表达特征与其功能和形态学特征联系起来。为确保这种分类方法分离出的细胞亚型具有相同的功能,他们提出在细胞分离和测序之前先确认它的形态和功能,使得细胞亚型标记物的识别更容易,分类更准确。Shekhar等用GFP转染双极细胞和Müller胶质细胞,FACS收集GFP阳性细胞,应用大规模平行单细胞转录组测序技术(Drop-seq)对约25 000个小鼠视网膜双极细胞进行分类,获得了15个细胞亚型。同时利用分子表达与细胞形态相匹配的方法验证了这种分类方法的准确性,并鉴定了数十种新的遗传标记物。单个视网膜亚型细胞遗传标记物的鉴定,有助于研究和绘制与特定视功能相关的靶点,使人们更深入地了解细胞功能及细胞异质性的来源,探究维持细胞多样性的基因网络。
另外,sc RNA-seq可用于罕见细胞类型的鉴别,如干细胞、前体细胞、肿瘤干细胞及循环肿瘤细胞等,以利于深入理解这些稀有细胞种群的生物学功能和特征。Kameishi等[40]发现兔角膜缘上皮旁群细胞与非旁群细胞相比具有干细胞样表型,应用GO(gene ontology)分析和RNA测序表明角膜缘上皮旁群细胞的间充质/内皮细胞标记物的表达较上皮细胞标记物显著增强,另外,单细胞qRT-PCR显示角膜缘上皮旁群细胞至少含有2种未成熟的细胞群,具有内皮样或间充质样表型,提示在角膜缘存在一种新型的角膜上皮干细胞,与传统的角膜上皮干细胞不同。目前,角膜缘上皮旁群细胞的研究促进了角膜上皮再生医学的进展,通过单细胞转录组的分析不仅仅鉴定了新的角膜上皮干细胞,同时也可以更好地理解干细胞或前体细胞在体内组织损伤修复过程中的作用。
2.2 sc RNA-seq对年龄相关性黄斑变性发病机制及治疗的研究 视网膜退行性病变,如视网膜色素变性(retinitis pigmentosa,RP)、年龄相关性黄斑变性(age-related macular degeneration,AMD),以光感受器细胞进行性损伤为特征,是发达国家视力丧失的主要原因[41]。先前关于AMD的遗传学研究认为可能的危险因素包括基因遗传、紫外线暴露、饮食习惯、心血管疾病、吸烟、饮酒等,其中基因遗传因素起非常重要的作用[42]。随着sc RNA-seq等基因组技术的研究进展,科学家将基因与发病机制相联系,进一步明确该疾病的发病机制。
Radeke的研究团队对68例捐献的眼组织进行单细胞转录组测序基因比较,其中包括31例正常眼,26例已确诊为AMD,11例被认为具有发展成为AMD的高危因素。研究发现50余个基因在AMD 细胞中高表达,其中前20个基因能够对AMD的诊断进行预测。在对RPE/脉络膜层的转录因子检测中,他们发现与凋亡相关的转录因子在干性AMD患者中呈高表达,而与血管生成相关的转录因子在湿性AMD患者中呈高表达。另外,他们还发现细胞介导的免疫反应是所有类型AMD的核心特征,由此推断AMD可能是一类具有共同的免疫反应过程的单一疾病。根据这些结果,针对核心免疫过程的药物可能成为AMD治疗的靶向药物。Morgan 等应用基因组技术比较了健康者和AMD患者的外周血及眼组织中基因表达和信号通路的不同,确定了特定危险基因与该疾病的关系,为实现特异性的靶点治疗提供科学依据。
2.3 sc RNA-seq对干细胞移植治疗视网膜退行性病变后基因单细胞转录组测序变化的评估
视网膜退行性病变尚无有效的治疗方法,干细胞或前体细胞移植是目前实验研究中具有潜能的治疗方法,可能终止或延缓视力丧失的进程。随着干细胞移植在视网膜退行性病变治疗方面研究的不断进展,其基因表达变化的分子机制需进一步研究。
Jones 等将人脑源性神经前体细胞(human neural progenitor cells,hNPC)移植到RCS (royal college of surgeons)视网膜退行性病变大鼠视网膜下,通过转录组测序分析获得实验组和对照组的视网膜中RNA数据,与假手术组RCS大鼠和 LE (long evans)大鼠对照。分析得出RCSsham 与LEsham有1215个基因差异性表达,RCShNPCs与 RCSsham 有283个基因差异性表达,比较这2组基因,发现68个逆表达基因(称为救援基因),包括Pdc、Rp1和 Cdc42ep5。该单细胞转录组测序研究结果为研究hNPC移植治疗视网膜退行性病变后的基因表达变化提供依据。在将来的干细胞移植治疗视网膜退行性病变的研究中,sc RNA-seq可用于增强或预测治疗的反应。
2.4 sc RNA-seq在眼部肿瘤的靶向治疗研究中的应用。
众所周知,肿瘤的分子特征和细胞特征可提示肿瘤细胞的来源,为靶向治疗提供依据。视网膜母细胞瘤是一种婴幼儿常见的眼内恶性肿瘤。1897年Wintersteiner曾尝试将视网膜母细胞瘤的细胞特征与特异性的视网膜细胞类型联系起来,认为光感受器细胞可能是肿瘤细胞的来源。还有人则认为其来源于前体细胞、胶质细胞或无长突细胞。关于肿瘤细胞的来源问题引起的争论大约持续了一个世纪。
如今,Mcevoy等对视网膜母细胞瘤患者及小鼠模型的肿瘤细胞进行单细胞基因表达阵列分析,显示在单一视网膜母细胞瘤的细胞中存在多种细胞类型特异性表达,主要包括光感受器细胞、视网膜前体细胞和含有无长突细胞与水平细胞的中间神经元,这种现象在正常视网膜发育过程中是不存在的,说明在肿瘤的发生过程中,控制视网膜发育和构建不同细胞类型的信号通路受到干扰。该研究指出从单细胞水平综合分析肿瘤的分子、细胞及生理学特征,发现神经递质受体、转运蛋白和生物合成酶在人视网膜母细胞瘤中表达,干扰这些通路可以减少肿瘤在体内外的生长。靶向神经递质儿茶酚胺和吲哚胺受体或下游成分在未来的单细胞转录组测序研究中可能成为视网膜母细胞瘤治疗的新靶点。
2.5 sc RNA-seq在眼外伤引起的基因表达变化中的应用
眼球爆炸伤在严重损坏视功能的同时还会发生基因的改变。Struebing等利用50 psi(1 psi=6.895 kPa)的高压空气波致54只小鼠眼球爆炸伤,5 d 后对视网膜RNA表达进行分析,约40%基因表达发生变化。通过机器学习算法,发现与天然免疫和获得性免疫相关的基因网络活化,伴随淋巴细胞入侵到视网膜内层,爆炸损伤还会引起视功能和神经节细胞的进行性丧失。
3总结与展望
随着分子生物学和生物信息学的不断发展,sc RNA-seq从技术和设备等多方面不断优化,涌现出一系列的商业化的测序平台,具有单次样本量大、耗时短、费用低、通量高等特点。sc RNA-seq可以从单个细胞的分子水平获得全基因组的表达谱,深入了解细胞个体间的差异,已广泛应用于干细胞的分化、胚胎及器官的发育、肿瘤的发生发展、神经系统及免疫系统等多个研究领域,并取得长足的进步。近年来,该单细胞转录组测序技术在眼科研究领域的应用也日渐增多。
sc RNA-seq可鉴别眼部细胞类型特异性标记物,有助于发现新的罕见细胞类型及细胞亚型,深入了解细胞间的个体差异。应用该技术分析AMD的发病机制,发现新的生物靶点和标记物,可能作为视网膜退行性疾病治疗的新方法。比较干细胞移植治疗视网膜退行性病变前后的基因表达变化,可用于增强或预测治疗的反应。通过该技术发现特异性标记物,可追溯视网膜母细胞瘤的肿瘤细胞来源,阐述肿瘤的发病机制,揭示肿瘤细胞的异质性,为肿瘤的预后评估及个体化治疗提供依据。sc RNA-seq与生物信息学技术的紧密结合,可为揭示细胞发育和分化过程中的基因调控网络提供强有力的检测方法。
目前,sc RNA-seq在眼科研究领域的应用仍较局限,随着该单细胞转录组测序技术的不断进步,在未来数年中可能迅速扩展到眼部相关疾病的研究中,如评估干细胞或前体细胞向眼组织的分化过程中的基因表达变化;探索干细胞眼内移植引起免疫排斥反应的发生机制;研究各种眼部肿瘤的发病机制、个体化治疗和预后的评估等。该技术将逐渐应用于临床,指导疾病诊断及治疗。