单细胞测序样本制备仪,基于Drop-seq技术*,完成高通量的单细胞mRNA 3’端测序。
已经对数千个线粒体基因组进行了测序,但可用的线粒体转录组相对较少。这可能很快就会改变。高通量RNA测序(RNA-Seq)技术使得生成大量线粒体转录组数据变得快速而廉价。在这里,我们探索RNA-Seq用于组装线粒体基因组和研究它们的表达模式的效用。
具体来说,西安大略大学的研究人员研究了Polytomella非光合作用绿藻的线粒体转录组,减少的线粒体基因组以及片段化的rRNA编码区,回文基因和线性染色体。端粒。从四种已知的Polytomella物种中分离全基因组RNA,然后进行Illumina配对末端测序,产生足够的线粒体衍生的读数,以容易地恢复几乎整个线粒体基因组序列。读取映射和覆盖统计数据还提供了对Polytomella线粒体转录结构的了解,揭示了多顺反子转录本以及端粒和回文基因的表达。
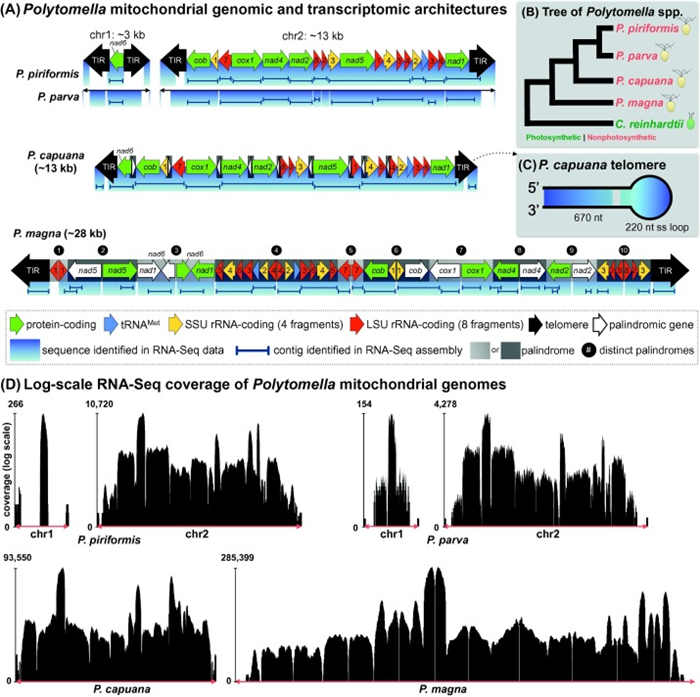
A.四种已知的Polytomella谱系的线粒体基因组图谱。所有四个基因组都由具有末端反向重复(TIR)端粒的线性染色体(chr)组成,并含有10个独特的基因,包括小的和大的亚基rRNA(SSU和LSU)基因,它们被分段并乱成4和分别是8个基因座。P. magna mtDNA含有10个回文重复序列(盒装为深色或浅灰色,并标有黑色圆圈),其中含有推定的功能性(绿色)和推定的非功能性基因拷贝(白色)。RNA-Seq数据中表示的区域以蓝色突出显示。从实体转录组装配鉴定的线粒体重叠群用实线显示。B.基于Smith等人的系统发育分析,Polytomella藻类和莱茵衣藻(Chlamydomonas reinhardtii)的树木。(2013)。C.来自P. capuana的单链(ss)发夹环端粒的转录。
该研究的主要目的是评估RNA-Seq用于恢复线粒体基因组序列的效用。在这方面,研究人员获得了成功:来自Polytomella物种的适量RNA-Seq数据很容易得到接近完整的线粒体基因组组装。Polytomella线粒体染色体的小尺寸和多顺反子转录组织无疑促进了它们从RNA数据中的有效回收。尽管如此,他们认为这里采用的方法可用于产生来自其他真核物种的细胞器基因组序列,包括那些mtDNA比Polytomella spp 更大的细胞。
RNA-Seq是一种用于研究线粒体遗传学的有前途的,具有成本效益的技术,但它确实存在缺陷。如图所示,其潜力之一是它可用于产生近乎完整的线粒体基因组序列,这在缺乏可用的mtDNA数据的情况下特别有用。